Zitierhinweis: Taubitz, Jan (2022) Entwicklung eines Open-Science-Indikators am Beispiel des FAIR Data Dashboards der Charité – Open Access Blog Berlin. DOI: https://doi.org/10.59350/nyts9-7q976
Dieser Beitrag zeigt am Beispiel des FAIR Data Dashboards der Charité — Universitätsmedizin Berlin, wie Open-Science-Indikatoren entstehen können. Hierzu greifen eine Reihe von selbst- und weiterentwickelten Datenanalyse- und Datenextraktions-Tools ineinander, um die FAIRness von in Datenrepositorien publizierten Forschungsdaten einer Forschungseinrichtung zu analysieren.
An wen richtet sich dieser Beitrag?
- An alle, die sich für Open Science Monitoring und die Entwicklung von Open-Science-Indikatoren interessieren.
- An alle, die sich mit FAIR Data Assessment beschäftigen — insbesondere mit einem automatischem FAIR Data Assessment auf institutioneller Ebene.
Was sind die FAIR Principles?
- 2016 veröffentlicht als FAIR Guiding Principles for scientific data management and stewardship
- Open-Data-Grundsätzen folgend sollen sie sicherstellen, dass Forschungsdaten findable, accessible, interoperable und reusable sind
- Der Fokus liegt auf der Maschinenlesbarkeit von (Meta-)Daten
Die Ausgangssituation
Unser Ziel war klar: Wir wollten im BUA-geförderten Projekt Open Science Dashboards einen Open-Science-Indikator entwickeln, der die Nachnutzbarkeit von Forschungsdaten von Forschenden der Charité anhand der FAIR Principles evaluiert.
Im ersten Schritt mussten wir herausfinden, welche Forschungsdaten überhaupt in öffentlich zugänglichen Datenrepositorien veröffentlicht sind. Hierzu wurde am BIH QUEST Center ein Workflow mit selbst- und weiterentwickelten Tools erstellt, um die in öffentlich zugänglichen Repositorien veröffentlichten Forschungsdaten von Angehörigen der Charité zu ermitteln. Der Workflow startet beim Journal-Artikel. Dieser Startpunkt der Analyse wurde gewählt, da wir nur Forschungsdaten analysieren wollten, die mit einem Forschungs-Output in Form eines Artikels verknüpft sind. Wir waren allerdings auch aus praktischen Gründen angeraten, beim Forschungsartikel zu starten. Für Forschungsartikel existieren eine Vielzahl bibliographischer Datenbanken, in denen nach Artikeln mit Charité-Affiliation gesucht werden kann. Für unser Vorhaben wurden beispielsweise die bibliographischen Meta-Datenbanken Web of Science und Embase verwendet. Für Forschungsdaten ist die Suche in Meta-Datenbanken anhand der Affiliation der Autor*innen bislang nicht zuverlässig möglich, was hauptsächlich daran liegt, dass in den existierenden Datenbanken viele Repositorien noch nicht indexiert werden.
Text Mining der Journal-Artikel mit ODDPub
Um aus der Publikationsliste zu ermitteln, welche Forschungsartikel mit veröffentlichten Forschungsdaten verknüpft sind, wurde das R-Package fulltext genutzt. Damit können Volltexte von abonnementbasierten Artikeln, die von mehreren großen Verlagen via API angeboten werden (z.B. Elsevier, Wiley oder Springer/Nature), sowie Volltexte von Open-Access-Artikeln über die Unpaywall API heruntergeladen werden. Die Volltexte wurden in einem nächsten Schritt mit dem ebenfalls selbstentwickelten Text-Mining-Algorithmus Open Data Detection in Publications (ODDPub) analysiert. ODDPub identifiziert die Data Statements in Artikeln und kennzeichnet diejenigen Publikationen, in denen Hinweise auf Daten-Repositorien und/oder Akzessions-Nummern von Forschungsdaten gegeben sind.

ODDPub detektiert zwar ziemlich präzise, ob der Artikel mit Forschungsdaten verknüpft ist. Das Tool zeigt jedoch nicht, mit welchen Forschungsdaten ein Artikel verknüpft ist und es kann nicht erkennen, ob die Forschungsdaten auch tatsächlich von den Autor*innen des Artikels erstellt wurden oder ob es sich um eine Nachnutzung von Forschungsdaten handelt, was wir in unserer Analyse nicht berücksichtigen wollten.
Manuelles Screening der Data Statements und Journal-Artikel mit Numbat
Um die durch ODDPub ermittelten Data Statements zu validieren, kommt ein weiteres Tool zum Einsatz. Es handelt sich um Numbat. Es ist ein Screening Tool, das ursprünglich für die Extraktion von Daten aus Primärquellen oder Artikeln beispielsweise für Systematic Reviews und andere Übersichtsarbeiten programmiert wurde. Mit einigen Anpassungen konnte es jedoch für unsere Zwecke genutzt werden. Durch einen manuellen Fragepfad kann mit Numbat zuverlässig ermittelt werden, ob es sich bei den im Artikel zitierten Forschungsdaten um selbsterstellte Forschungsdaten handelt und welche IDs diese Forschungsdaten haben. Der Nachteil von Numbat ist sicherlich, dass es einen hohen zeitlichen Aufwand verursacht, da es auf einem manuellen Screening basiert. Der ODDPub- und Numbat-Workflow ist ausführlich in einem Protokoll auf protocols.io beschrieben.
Am Ende von Numbat und des gesamten Workflows, der mit der Publikationsliste startet, steht eine Liste mit Globally Unique Identifiers (GUIDs: URLs oder DOIs) von in Repositorien veröffentlichten und mit Charité-Autor*innen verknüpften Datensätzen.
FAIR Assessment mit F-UJI
Die IDs können nun auf ihre Nachnutzbarkeit nach den FAIR Principles überprüft werden. Hierzu gibt es eine Reihe von Assessment-Tools, die die Forschungsdaten-IDs als Input nehmen und Metadaten über die Landing Pages der Repositorien und/oder über DataCite abfragen. Mittels der abgefragten Metadaten kann die FAIRness evaluiert werden. Als zuverlässigstes Tool mit der besten Dokumentation und gutem Support hat sich F-UJI herausgestellt, das im Rahmen des FAIRsFAIR-Projekt entstanden ist und nun im FAIR-IMPACT-Projekt weiterentwickelt wird. Es gibt eine Reihe von Tools, die ähnlich funktionieren und vielversprechend sind. Hierzu gehören der FAIR Evaluator oder auch FAIR Enough. F-UJI (und die anderen Tools) sind allerdings für die Analyse einzelner Forschungsdaten konzipiert. Um unsere Liste mit Forschungsdaten abfragen zu können, musste F-UJI zunächst lokal installiert werden. Anschließend konnte über eine API unsere Liste mit den IDs abgefragt werden:
# Function to query local F-UJI server
fuji_local_server <- function(rd_id){
headers = c(
`accept` = "application/json",
`Content-Type` = "application/json")
data <- list(metadata_service_endpoint = "",
metadata_service_type = "oai_pmh",
object_identifier = rd_id,
test_debug = TRUE,
use_datacite = TRUE)
res <- httr::POST(url = "http://localhost:1071/fuji/api/v1/evaluate", httr::add_headers(.headers = headers), body = data, encode = "json")
fuji_local_parsed <- content(res)
return(fuji_local_parsed)
}
# Query large set of research data ids
fuji_local_list <- map(charite_rd_2020_guid, fuji_local_server)Der Output von F-UJI ist eine JSON-Datei mit dem FAIR Assessment. Das FAIR Assessment von F-UJI basiert auf den FAIRsFAIR-Metriken, die die abstrakten FAIR Principles zunächst in überprüfbare Metriken und anschließend in durchführbare Tests übertragen.
Der F-UJI-Output wurde noch mit Informationen über die Repositorien angereichert, die re3data (Registry of Research Data Repositories) bereitstellt. Anschließend wurde der Output statistisch analysiert.
Hier der gesamte Workflow in einem High Level Flowchart:
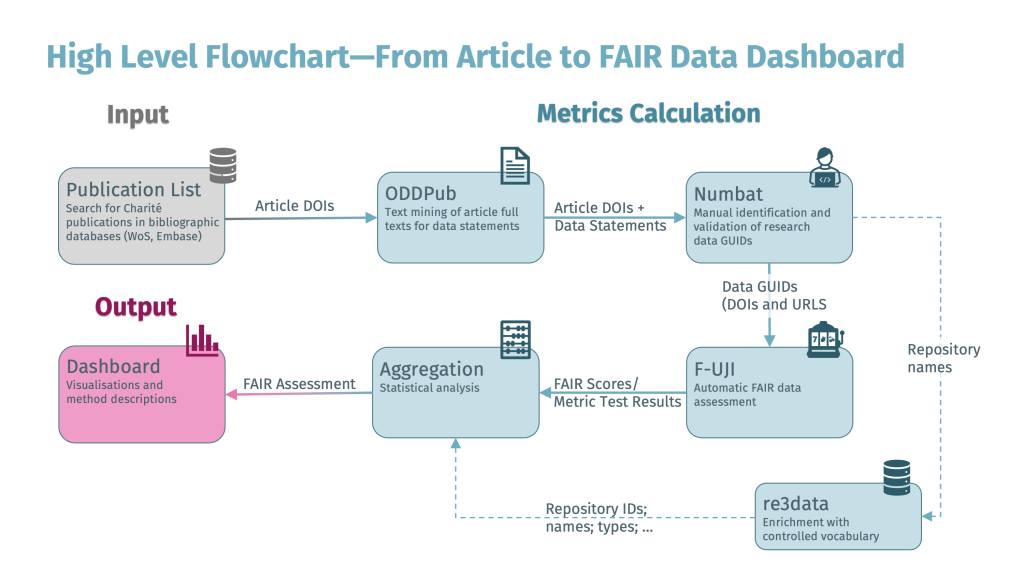
Präsentation der Ergebnisse in einem Dashboard
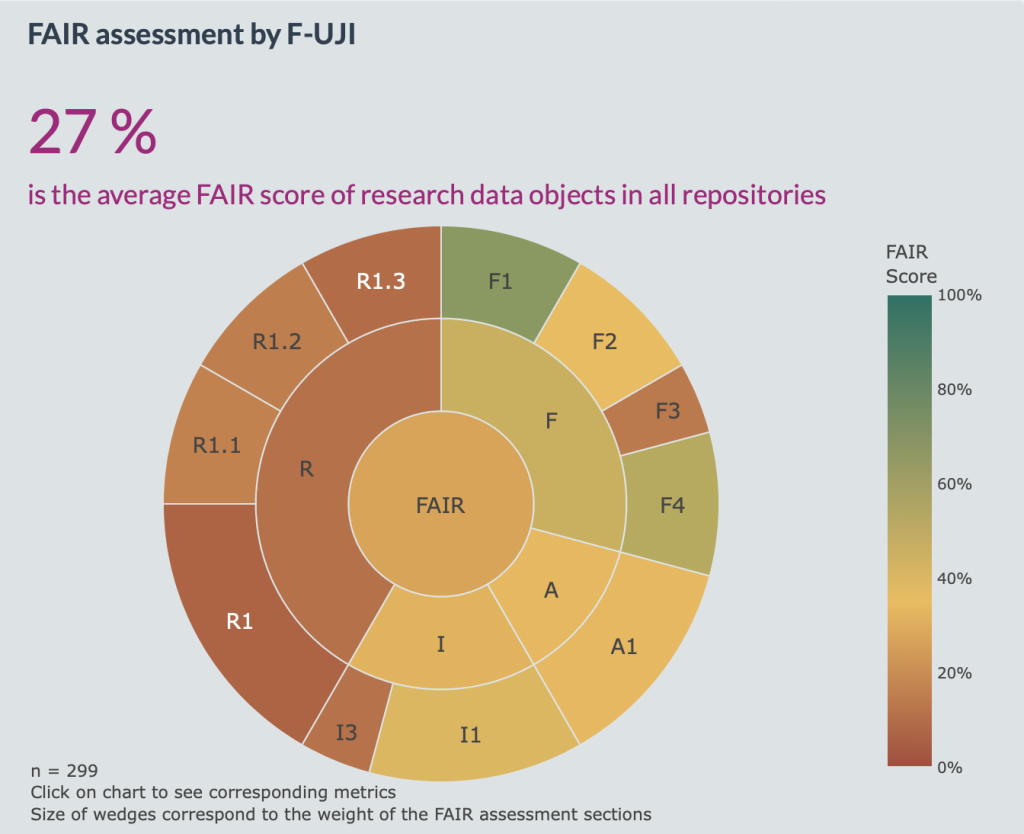
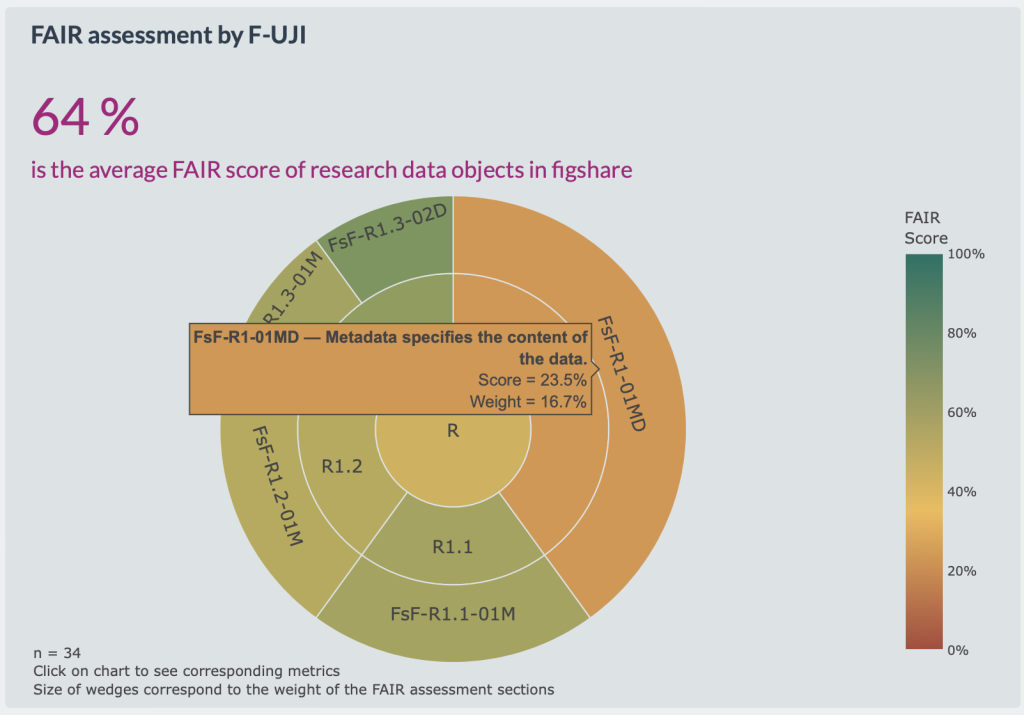
Das Ergebnis wurde als Teil des Charité Dashboards for Responsible Research veröffentlicht. Im Zentrum steht ein FAIR Score, der die prozentuale Erfüllung der FAIRsFAIR-Metriken abbildet.
Präsentiert werden die Ergebnisse u.a. in einem Sunburst-Chart, der auf einen Blick die Erfüllung und die Gewichtung der einzelnen FAIR Principles darstellt. Das F1-Prinzip („(Meta)data are assigned a globally unique and persistent identifier“) ist beispielsweise mit 8,3% gewichtet, wohingegen das R1-Prinzip („(Meta)data are richly described with a plurality of accurate and relevant attributes“) mit 16,7% in die Gesamtwertung einfließt.
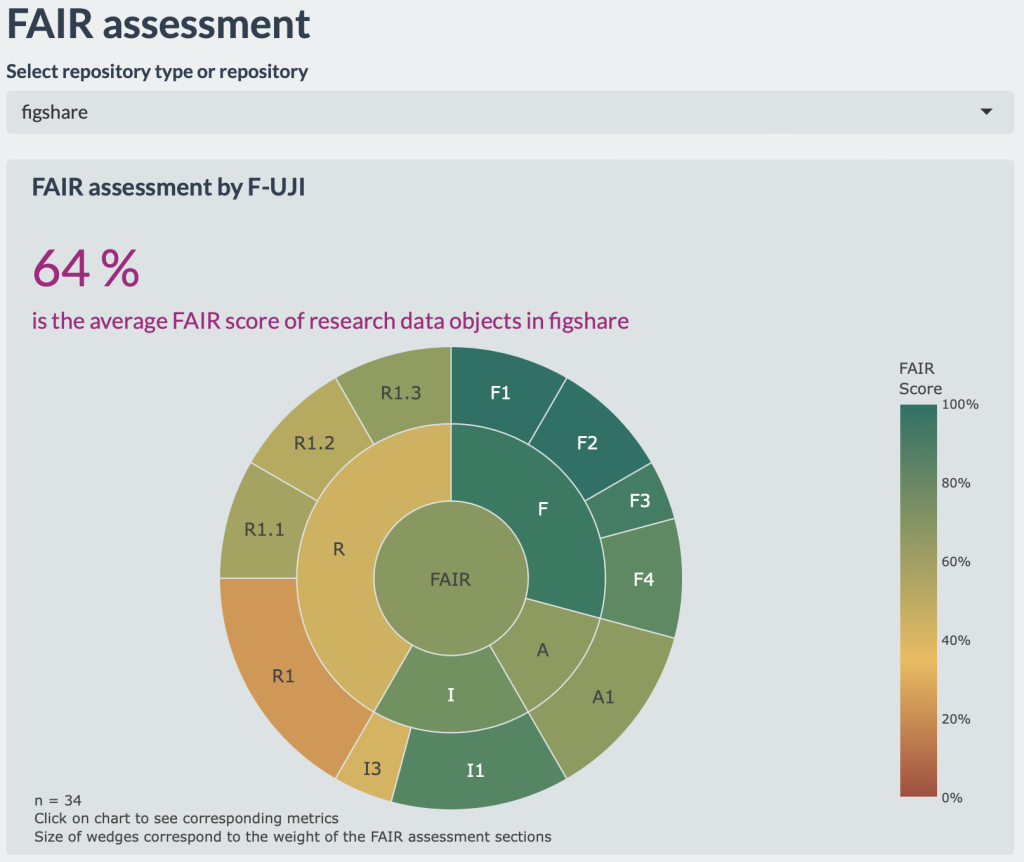
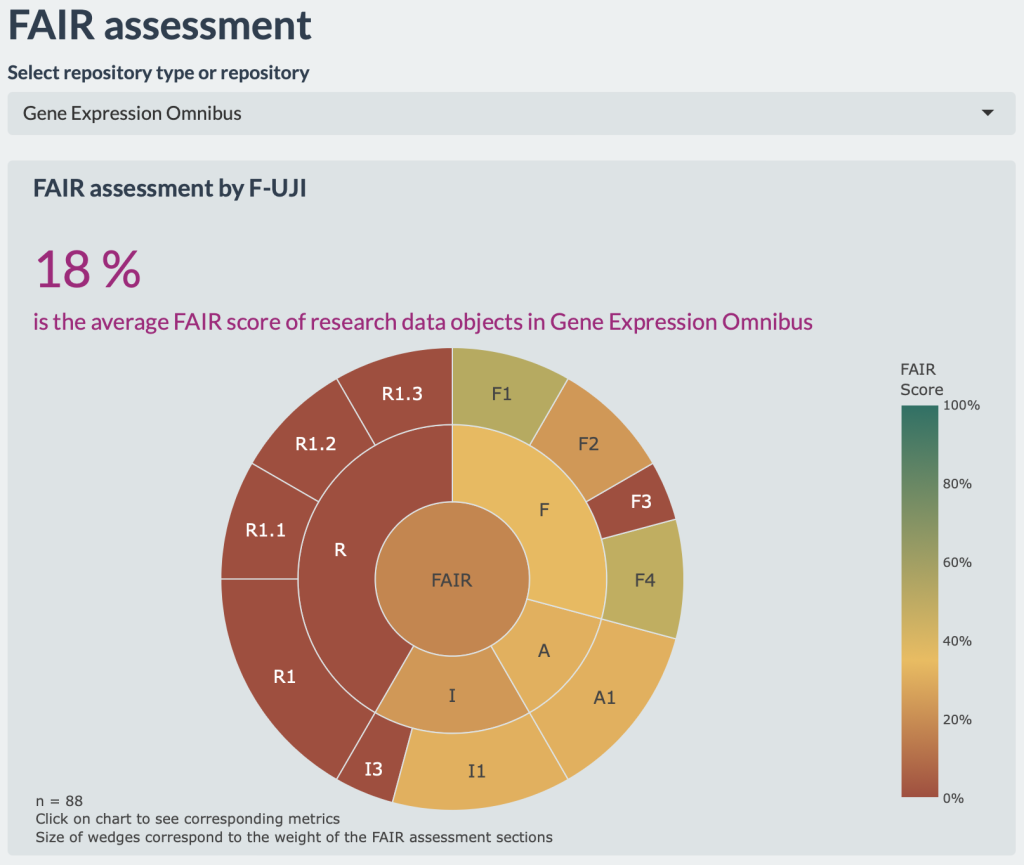
Die Ergebnisse der Evaluation können nach Repositoriums-Typ (fachspezifisch oder allgemein) sowie nach den einzelnen Repositorien gefiltert werden. So wird deutlich, dass allgemeine Repositorien wie Figshare oder Zenodo deutlich besser abschneiden als beispielsweise die an der Charité intensiv genutzten fachspezifischen Repositorien des U.S. National Center for Biotechnology Information (NCBI).
Was haben wir durch das FAIR-Assessment gewonnen?
Mit dem FAIR Score können wir die Nachnutzbarkeit von Forschungsdaten auf institutioneller Ebene bewerten. Unseres Wissens gibt es weltweit keine andere Einrichtung, die die FAIRness der von ihren Forschenden publizierten Forschungsdaten evaluiert und offenlegt.
Ein weiterer Vorteil dieser Darstellung ist, dass durch das FAIR Assessment und dessen Visualisierung die FAIR Principles nachvollziehbarer werden und somit Wissen über FAIR Data vermittelt werden kann. Das ist nötig, da unter der Oberfläche des eingängigen Akronyms FAIR die FAIR Principles schnell sehr technisch werden. Das FAIR Data Dashboard zeigt die hinter den FAIR Principles liegenden deutlich konkreteren FAIRsFAIR-Metriken, was Nutzer*innen des Dashboards über den Aufbau eines FAIR Assessments informiert. Neben dem wissensvermittelnden Aspekt gewinnen wir Informationen über die von an der Charité Forschenden verwendeten Datenrepositorien und deren FAIRness, die für Beratungen und Interventionen eingesetzt werden können.
Der FAIR Score ist zunächst nur eine Nummer, der die Ergebnisse verschiedener Metadaten-Tests zusammenfasst. Ob sich der FAIR Score zur Beschreibung der Nachnutzbarkeit von Forschungsdaten etabliert oder ob sich eine andere (anschaulichere?) Skala durchsetzt, wird sich zeigen.
Eine wichtige Einschränkung ist, dass durch das FAIR Assessment ausschließlich die Metadaten der Datensätze untersucht werden. Das heißt, die Qualität der den Forschungsdaten zugrundeliegende Forschung, die tatsächliche Reproduzierbarkeit der Analysen und die Vollständigkeit der Daten kann mit den automatischen Tools nicht überprüft werden. Das ist ein Aspekt, der auch andere Open-Science-Metriken betrifft: Ein grüner oder goldener Open-Access-Status trifft eine Aussage zur Offenheit des Forschungsartikels, aber nicht zu seiner Originalität oder methodischen Genauigkeit.
Das FAIR Data Dashboard und der FAIR Score ist ein Beispiel, wie Open-Science-Indikatoren durch den Einsatz von selbstentwickelten Tools sowie die Nutzung und Weiterentwicklung bestehender Tools entstehen können. Wer Interesse an der Entwicklung eigener Open-Science-Indikatoren hat, der sollte sich unseren Call for Participation ansehen:

